ProMode-Elastic :
二面角を独立変数とする弾性ネットワークモデルによるPDBデータの基準振動解析データベース
ProMode-Elasticは、
PDBにある立体構造データに対して行った基準振動解析の結果を公開しているデータベースです。計算には、われわれが開発したプログラムPDBETA (
Elastic-network-model
based normal mode analysis in
Torsional
Angle space for
PDB
data)
を使用しています。PDBETAは、二面角を独立変数とする弾性ネットワークモデルによる基準振動解析プログラムです。二面角を独立変数に採用することで、PDBデータの
すべての原子を考慮しても、分子構造を比較的少ない自由度で記述することができ、また、弾性ネットワークモデルを採用したことで、タンパク質分子のみならず、
DNA、RNA、リガンドなどの分子を取り入れた計算が可能になりました(ただし、水素原子と水分子については、変数の数を抑制するため、現時点では除外して
います)。
=> タンパク質立体構造の基準振動解析については、
こ
ちら をご覧ください。
それぞれのタンパク質のページでは、タンパク質分子の揺らぎを
Molmil
分子ビューワーによってアニメーションや原子の変位ベクトル図で観察することができます。静止画だけからは得られないタンパク質の動的構造に関す
る知見が、皆様の研究のお役に立つことを願っております。
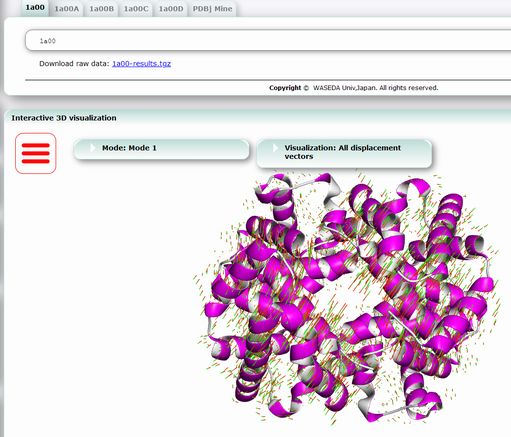
ProMode-Elastic の画像例(1)
変位ベクトル図およびアニメーション
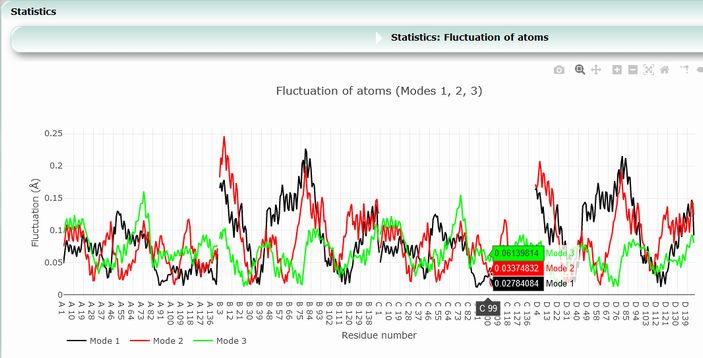
ProMode-Elastic の画像例(2)
Mode 1, 2, 3 における各残基のゆらぎの大きさ
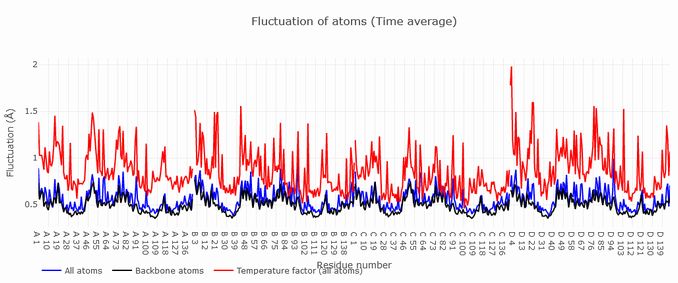
ProMode-Elastic の画像例(3)
各残基のゆらぎの大きさの時間平均
参考文献
- Hiroshi Wako and Shigeru Endo, "Normal mode analysis as a method to
derive protein dynamics information from the Protein Data Bank."
Biophys. Rev. Vol.9, 877-893 (2017) [doi:10.1007/s12551-017-0330-2]
- Hiroshi Wako and Shigeru Endo, "Normal mode analysis based on an
elastic network model for biomolecules in the Protein Data Bank, which
uses dihedral angles as independent variables." Comp. Biol. Chem. Vol.
44, 22-30 (2013) [doi:10.1016/j.compbiolchem.2013.02.006]
- Hiroshi Wako and Shigeru Endo, "Ligand-induced conformational
change of a protein reproduced by a linear combination of displacement
vectors obtained from normal mode analysis." Biophys. Chem. Vol. 159,
257-266 (2011) [doi:10.1016/j.bpc.2011.07.004]
【PDBj 内の関連ページ】
ページの最後に PDB ID の入力欄があり、そこから各タンパク質のページに進むことができます。
複数鎖からなる系では、複数鎖全体での基準振動解析および各鎖個別の基準振動解析
結果を見ることができます。各タンパク質の左上に、それらを選択するタグが表示されます。
【外部リンク】ProMode-Elastic とは何か
ProModeの概略が記されています。このページとほぼ同じ内容です。
ProMode-Elastic の使用方法などについて記されています。
このWEBサイトでも同様の説明を見ることができます。 ==>>> こ
ちら
【外部リンク】プログラム PDBETA
のダウンロード
プログラム PDBETA をダウンロードすることができます。