ProMode Elastic からの出力ファイルの利用の仕方
ProMode-Elastic では、各タンパク質について、計算結果をダウンロードできる。たとえば、PDB ID 1a00 に対して
と表示されるので、これをダウンロードする。この圧縮ファイルには以下のファイルが含まれている。なお、ファイル名のうち、xxxxは PDB ID
を、_n. はモード番号(1, 2, ..., 10)を表している。
2.1 xxxx-min.pdb
PDBフォーマットの原子座標データ。基準振動解析は、本来、エネルギー極小構造に対して行うものであるが、Elastic Network model
では、PDBの構造をエネルギー極小構造とみなし、計算を行っている。
2.2 xxxx.flcatmA
原子の揺らぎの大きさを各残基ごとに平均をとった値。単位はÅ。
[出力例]
# Normal mode analysis: PDB1bh9_AB
# Time average of atomic fluctuations (in unit of Angstroms).
# Average atomic displacements= 1.0A
# No. of residues= 136
Residue All atoms
Backbone atoms CA atom
LEU A 31
1.04354
0.90098
0.76359
1.45504
1.46276 1.45855
PHE A 32
0.71084
0.63711
0.53538
1.40053
1.41837 1.42348
SER A 33
0.84845
0.73939
0.60199
1.36845
1.36388 1.36920
LYS A 34
1.29700
0.72118
0.57441
1.41037
1.36967 1.36656
(中略)
ILE B 200
0.86540
0.69329
0.57963
1.32263
1.32049 1.32520
PRO B 201
0.91068
0.84586
0.71196
1.38792
1.39062 1.38355
1234567890123456789012345678901234567890123456789012345678901234567890123456789012345678901234567890
① ② ③ ④ ⑤ ⑥
カラーの文字行は参考のために付け加えたもので、実際には出力されない。
各残基ごとに、残基名、鎖名、残基番号に続いて6つの数字が出力されている。それぞれ次のような値である。
① 残基内のすべての原子の揺らぎを平均した値
② 残基内の主鎖原子(注1)の揺らぎを平均した値
③ Cα原子の揺らぎの値(ただし、DNAでは、C4’原子の揺らぎの値)
④ 温度因子から求めた残基内のすべての原子の揺らぎ(注2)を平均した値
⑤ 温度因子から求めた残基内の主鎖原子の揺らぎを平均した値
⑥ 温度因子から求めたCα原子の揺らぎの値(ただし、DNAでは、C4’原子の揺らぎの値)
(注1)主鎖原子として、N、Cα、Cβ、C、Oを考えている。
(注2)PDBに与えられている温度因子 から原子の揺らぎは として求めた。
2.3 xxxx.flcangA
各二面角の揺らぎの大きさ。単位は度。
[出力例]
# Normal mode analysis: PDB1bh9_AB
# Time avarage of dihedral angle fluctuations.
# Average atomic displacements= 1.0A
# No. of residues= 136
# Root Mean Square Deviation of Dihedral Angles (in unit of degrees)
Residue
Phi
Psi
Omega
Chi1
Chi2
Chi3 ・・・
LEU A 31 5
0.00000 28.46658
19.66447 36.93524 43.50697
PHE A 32 5
26.22306 24.72205
16.02092 13.64694 25.29796
SER A 33 4
36.75111 29.68622
23.32820 59.64042
LYS A 34 7
34.09119 28.84911
20.71714 40.10385
67.64747 100.48421 ・・・
(中略)
GLN B 199 6
30.90751 26.84232
18.67794 47.96300
30.51616 67.12095
ILE B 200 5
34.41804 18.68154
20.80474 26.76434 57.01173
PRO B 201 2
0.00000 39.07812
1234567890123456789012345678901234567890123456789012345678901234567890123456789012345678901234567890
カラーの文字は参考のために付け加えたもので、実際には出力されない。
各残基ごとに、残基名、鎖名、残基番号に続いて、その残基内の二面角の数が与えられ、二面角の揺らぎの大きさが、φ、ψ、ω、χ1、χ2、χ3 …の順に与え
られている。
2.4 xxxx.crratmA
原子の揺らぎの相関。
[出力例]
# Normal mode analysis: PDB1bh9_AB
# Average atomic displacements= 1.0A
# Time average of correlations between atomic fluctuations
(non-normalized).
# No. of atoms= 134
1234567890123456789012345678901234567890123456789012345678901234567890123456789012345678901234567890
LEU A 31 PHE A
32 SER A
33 LYS A
34 GLU A 35
CA
CA
CA
CA
LEU A 31 CA 0.58307E+00
PHE A 32 CA
0.17856E+00 0.28664E+00
SER A 33 CA
0.60482E-01 0.14210E+00 0.36240E+00
LYS A 34 CA
0.36273E-01 0.61709E-01
0.17023E+00 0.32994E+00
(中略)
GLN B 199 CA 0.16043E-01
-0.17150E-02 -0.11016E-01 -0.99436E-02
-0.18139E-01
ILE B 200 CA 0.78266E-02
-0.46050E-02 -0.13576E-01 -0.17480E-01
-0.21192E-01
PRO B 201 CA -0.24784E-02
-0.13074E-01 -0.20272E-01 -0.26459E-01
-0.30231E-01
1234567890123456789012345678901234567890123456789012345678901234567890123456789012345678901234567890
# Time average of correlations between atomic fluctuations (normalized).
# No. of atoms= 134
LEU A 31 PHE A 32 SER A
33 LYS A 34 GLU A 35 LEU
A 36
CA
CA
CA
CA
CA
CA
LEU A 31 CA 1.00000
PHE A 32 CA
0.43678 1.00000
SER A 33 CA
0.13158 0.44089 1.00000
LYS A 34 CA
0.08270 0.20066
0.49228 1.00000
(中略)
GLN B 199 CA
0.04511 -0.00442
-0.03108 -0.02590
-0.04695 -0.07271
ILE B 200 CA
0.02596 -0.01401
-0.04517 -0.05370
-0.06470 -0.08629
PRO B 201 CA
-0.00669 -0.03239
-0.05492 -0.06617
-0.07514 -0.08636
1234567890123456789012345678901234567890123456789012345678901234567890123456789012345678901234567890
カラーの文字は参考のために付け加えたもので、実際には出力されない。
2つの部分からなる。
前半部分では、縦、横に示された原子対i, jの揺らぎ 、 の規格化されていない相関の時間平均、すなわち、 が与えられる。ここで < >
は、すべてのモードについて時間平均をとることを意味する
後半部分では、規格化された相関、すなわち、 が与えられる。値は、-1.0から1.0となる。
2.5 xxxx.crrangA
二面角の揺らぎの相関。
[出力例]
# Normal mode analysis: PDB1bh9_AB
# Average atomic displacements= 1.0A
# Time average of correlations between dihedral angle fluctuations
(non-normalized).
# No. of dihedral angles= 126
1234567890123456789012345678901234567890123456789012345678901234567890123456789012345678901234567890
Non-normalized PHE A 32
SER A 33 LYS A 34 GLU
A 35 LEU A 36 ARG A
37
correlation
PHI
PHI
PHI
PHI
PHI
PHI
PHE A 32 PHI 0.68765E+03
SER A 33 PHI 0.95230E+02 0.13506E+04
LYS A 34 PHI -0.20845E+02 -0.12682E+03 0.11622E+04
GLU A 35 PHI -0.15646E+02 0.16318E+01
0.39971E+02 0.96582E+03
(中略)
GLY B 198 PHI 0.30166E+01 -0.15392E+00 0.25792E+01
-0.63852E+01 0.35279E+01 0.93945E+00
GLN B 199 PHI -0.30212E+00 0.22415E+00
-0.74516E+00 0.18983E+01 -0.53020E+00 -0.22954E+00
ILE B 200 PHI -0.16864E+01 0.41431E+00
-0.10480E+00 0.74331E+00 -0.60455E+00 -0.20844E+01
1234567890123456789012345678901234567890123456789012345678901234567890123456789012345678901234567890
# Time average of correlations between dihedral angle fluctuations
(normalized).
# No. of dihedral angles= 126
1234567890123456789012345678901234567890123456789012345678901234567890123456789012345678901234567890
Normalized PHE A
32 SER A 33 LYS A
34 GLU A 35 LEU A
36 ARG A 37
correlation
PHI
PHI
PHI
PHI
PHI
PHI
PHE A 32 PHI 1.00000
SER A 33 PHI
0.09881 1.00000
LYS A 34 PHI
-0.02332 -0.10122
1.00000
GLU A 35 PHI
-0.01920
0.00143
0.03773 1.00000
(中略)
GLY B 198 PHI
0.00157 -0.00007
0.00116 -0.00347
0.00200 0.00028
GLN B 199 PHI
-0.00028 0.00018
-0.00060 0.00185
-0.00054 -0.00012
ILE B 200 PHI
-0.00141 0.00030
-0.00008 0.00065
-0.00055 -0.00102
1234567890123456789012345678901234567890123456789012345678901234567890123456789012345678901234567890
カラーの文字行は参考のために付け加えたもので、実際には出力されない。
2つの部分からなる。
前半部分では、縦、横に示された二面角の対i, jの揺らぎ 、 の規格化されていない相関の時間平均、すなわち、 が与えられる。ここで <
> は、すべてのモードについて時間平均をとることを意味する
後半部分では、規格化された相関、すなわち、 が与えられる。値は、-1.0から1.0となる。
2.6 xxxx_n.flcatmE
モード番号 n の基準振動での各原子の変位ベクトル。
[出力例]
# Normal mode analysis: PDB1bh9_AB
# Average atomic displacements= 1.0A
# No. of atoms= 1074
# 1-th mode (Frequency= 54.093 (/cm),
Period= 6.16791E-13 (sec) )
Atomic coordinate
Displacement vector
x
y
z
x
y z abs. value
1 N LEU A
31 35.269 -1.755
31.866 0.160 0.469
-0.188 0.530
2 CA LEU A
31 33.817 -1.355
31.863 0.157 0.459
-0.183 0.518
3 CB LEU A
31 33.747 0.148
31.895 0.334 0.474
-0.496 0.763
(中略)
1072 C PRO B 201
32.102 17.925 22.627
-0.173 0.236 0.129 0.319
1073 O PRO B 201
32.928 17.384 21.850
-0.166 0.234 0.138 0.318
1074 OXT PRO B 201 32.439
18.524 23.670 -0.181
0.243 0.128 0.329
1234567890123456789012345678901234567890123456789012345678901234567890123456789012345678901234567890
① ② ③ ④ ⑤ ⑥ ⑦
カラーの文字行は参考のために付け加えたもので、実際には出力されない。
各原子ごとに、原子の通し番号、原子の種類、属する残基名、鎖名、残基番号に続き、PDBのx, y, z の座標値が①、②、③に、変位ベクトルのx,
y, zとその絶対値がそれぞれ④、⑤、⑥、⑦に与えられている。
2.7 xxxx_n.flcangE
モード番号 n の基準振動での各二面角の変位。
[出力例]
# Normal mode analysisten PDB1bh9_AB
# Average atomic displacements= 1.0A
# 1-th mode (Frequency= 54.093 (/cm),
Period= 6.16791E-13 (sec) )
# Displacements of dihedral angles (in unit of degrees)
# No. of residues= 136
Residue
Phi
Psi
Omega
Chi1
Chi2
Chi3
LEU A 31 5
0.00000
0.43439
0.37093 0.52906
-0.18606
PHE A 32 5
-0.40392
1.42156 0.77656
-0.14650 0.07097
SER A 33 4
-1.35308 -0.57450
0.15308 0.07695
LYS A 34 7
0.10480 0.57011
-0.12007 -0.90313
0.22883 -0.31029
(中略)
GLY B 198 3
-0.08340 0.29035
-0.15886
GLN B 199 6
-0.20385 -0.35576
-0.20143 0.06844
-0.15228 0.12705
ILE B 200 5
0.64593 0.11304
-0.05599 -0.13481
0.06314
PRO B 201 2
0.00000 -0.09151
1234567890123456789012345678901234567890123456789012345678901234567890123456789012345678901234567890
カラーの文字は参考のために付け加えたもので、実際には出力されない。
2.8 xxxx_n-anm.PDB
モード番号 n の基準振動の jV用のアニメーション・データ。jmol でも使うことができる。
[出力例]
REMARK Normal mode analysis: PDB1bh9_AB
REMARK Average atomic displacements= 1.0A
REMARK
REMARK 1-th mode
(Frequency= 54.093 (/cm), Period= 6.16791E-13 (sec) )
MODEL 1
1234567890123456789012345678901234567890123456789012345678901234567890123456789012345678901234567890
ATOM 1 N LEU A
31 35.557 -1.497 32.096
1.00 0.00
ATOM 2 CA LEU A
31 34.101 -1.113 32.103
1.00 0.00
ATOM 3 C LEU A
31 33.321 -1.674 30.934
1.00 0.00
(中略)
ATOM 1073 CG PRO B
201 29.617 15.698 22.914
1.00 0.00
ATOM 1074 CD PRO B
201 29.336 15.718 21.441
1.00 0.00
ATOM 1075 OXT PRO B 201
32.205 18.469 23.506 1.00 0.00
ENDMDL
MODEL 2
ATOM 1 N LEU A
31 35.538 -1.513 32.078
1.00 0.00
ATOM 2 CA LEU A
31 34.082 -1.129 32.085
1.00 0.00
ATOM 3 C LEU A
31 33.303 -1.688 30.915
1.00 0.00
(中略)
ATOM 1073 CG PRO B
201 29.779 15.794 23.207
1.00 0.00
ATOM 1074 CD PRO B
201 29.448 15.858 21.746
1.00 0.00
ATOM 1075 OXT PRO B 201
32.450 18.481 23.781 1.00 0.00
ENDMDL
MODEL 11
ATOM 1 N LEU A
31 35.107 -1.854 31.899
1.00 0.00
ATOM 2 CA LEU A
31 33.668 -1.411 31.877
1.00 0.00
ATOM 3 C LEU A
31 32.908 -1.882 30.657
1.00 0.00
ATOM 4 O LEU A
31 31.800 -2.386 30.794
1.00 0.00
(中略)
ATOM 1072 CB PRO B
201 29.740 17.252 23.537
1.00 0.00
ATOM 1073 CG PRO B
201 29.774 15.791 23.211
1.00 0.00
ATOM 1074 CD PRO B
201 29.441 15.856 21.750
1.00 0.00
ATOM 1075 OXT PRO B 201
32.446 18.476 23.786 1.00 0.00
ENDMDL
END
カラーの文字行は参考のために付け加えたもので、実際には出力されない。
データはPDBデータと同じ様式である。アニメーションのフレーム数nfに相当するだけの立体構造データがそれぞれ、MODEL行とENDMDL行に挟まれ
て出力される。
[jVでアニメーションを見る]
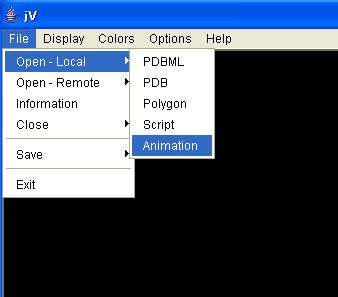
① jVを起動し、このファイルを読み込む。 [File] → [Open-Local] → [Animation]
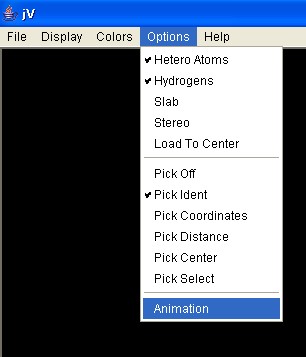
② アニメーションをスタートさせる。 [Options] → [Animation]
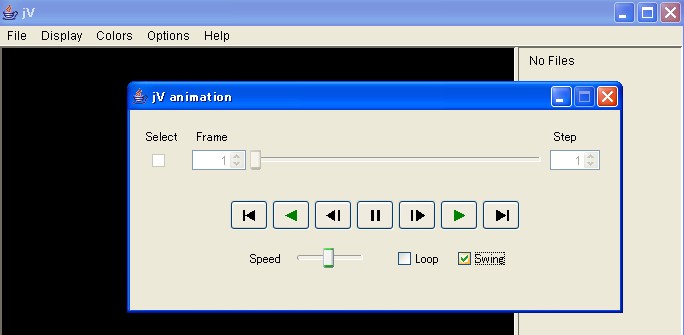
③ アニメーションの操作盤が現れるので、swingにチェックを入れ、再生ボタン▷をクリックする。
(swingにチェックを入れることで、データ内の立体構造が 1, 2, …, nf-1, nf, nf-1, …, 2,
1の順に表示され、これが繰り返される。ここで nf は、データ内に含まれる立体構造の総数で、ここでは 11 に設定している。)
[jmolでアニメーションを見る]
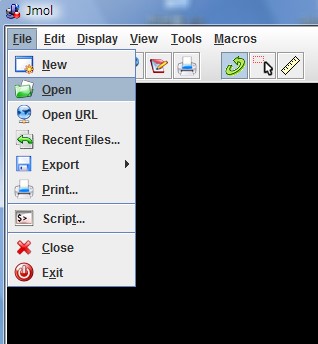
① jmolを起動し、このファイルを読み込む。 [File] → [Open]
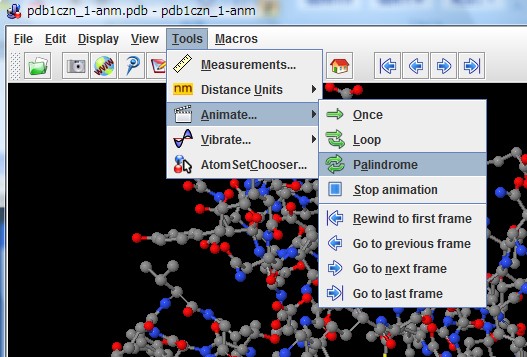
② アニメーションをスタートさせる。 [Tools] → [Animate] → [Palindrome]
(Palindromeを選択することで、データ内の立体構造が 1, 2, …, nf-1, nf, nf-1, …, 2,
1の順に表示され、これが繰り返される。ここで nf は、データ内に含まれる立体構造の総数であり、ここでは 11 に設定している。)
[アニメーションGIFを作る]
xxxx_n-anm.PDB には、PDBフォーマットの <MODEL> <ENDMDL>
タグを使って11個の構造が入っている。上記の [jV でアニメーションを見る] に従ってこのファイルを読み込み、animation
を起動すると、操作パネルが現れ、animation を実行できるが、11個の構造を個別に表示することもできる。それを [FIle] →
[Save] → [JPG] で画像として出力する。例えば、それらのファイル名を img001.jpg, img002.jpg, ...,
img011.jpg とする。出力したファイルのうち、img011.jpg
を除くファイルについてコピーを作り、ファイル名を img010.jpg → img012.jpg, img009.jpg →
img013.jpg, ... , img001.jpg → img021.jpg
とする。これらをアニメーションGIF作成のソフトを使ってアニメーションGIFを作成することができる。
たとえば、アニメーションGIFの作成ソフトには Giam がある。「窓の杜」からダウンロードする場合には こちらから。
2.9 xxxx_n-vec.xml
モード番号 n の基準振動の変位ベクトルを jV で描画するためのデータ。XML形式で出力される。
[出力例]
<?xml version="1.0"?>
<polygon>
<vertices id_numbers="use">
<vertex id="1" image="35.269 -1.755
31.866 0.160 0.469
-0.188 0 128
0"></vertex>
<vertex id="2" image="35.748 -0.347
31.302 0.160 0.469
-0.188 255 0
0"></vertex>
<vertex id="3" image="33.817 -1.355
31.863 0.157 0.459
-0.183 0 128
0"></vertex>
(中略)
<vertex id="2146" image="32.430 18.086
22.263 -0.166 0.234
0.138 255 0 0">
</vertex>
<vertex id="2147" image="32.439 18.524
23.670 -0.181 0.243
0.128 0 128 0">
</vertex>
<vertex id="2148" image="31.895 19.254
24.053 -0.181 0.243
0.128 255 0 0">
</vertex>
</vertices>
<line_array count="1074" width="2.0">
<line id="1" vertex="1 2"/>
<line id="2" vertex="3 4"/>
<line id="3" vertex="5 6"/>
(中略)
<line id="1072" vertex="2143 2144"/>
<line id="1073" vertex="2145 2146"/>
<line id="1074" vertex="2147 2148"/>
</line_array>
</polygon>
[jVで変位ベクトルを描画する]
① jVを起動し、対応するPDBデータを読み込む。 [File] → [Open-Local] → [PDB]
② 変位ベクトル描画用ファイルを読み込む。 [File] → [Open-Local] → [Polygon]
(注意)PDBデータの構造と変位ベクトルとは同じ座標系になくてはならない。①、②の操作
中、画面に表示された構造や変位ベクトルに対して、回転、並進、拡大・縮小といった操作をすると、両者の間にずれが生じる。ちょっとしたマウス操作で、思
いもかけずこうした操作を行ってしまうことがあるので注意する必要がある。
①、②の操作を繰り返して、複数の変位ベクトル描画用ファイルを読み込むことができる。複数のモードの変位ベクトルを読み込んだ場合、jV
の右側のパネルに、それぞれのモードの変位ベクトルを「表示する」、「非表示にする」ためのチェックボックスが現れるので、それを利用してモードごとに表示・非表示を後か
ら指定できる。
また、変位ベクトルにはmainchainという属性が設定してあり、主鎖原子は mainchain=1、側鎖原子は mainchain=0
である。これを利用して、主鎖原子と側鎖原子の変位ベクトルの表示・非表示を制御することができる。
[例1] すべての変位ベクトルが表示されているとき(デフォルト)、jV のコマンド
selectvertex mainchain=0
displayvertex off
を打ち込むことで、側鎖原子の変位ベクトルを非表示にすることができる。
[例2] 変位ベクトルがまったく表示されていないとき、jV のコマンド
selectvertex mainchain=1
displayvertex on
を打ち込むことで、主鎖原子の変位ベクトルだけを表示することができる。
[変位ベクトルの表示例]
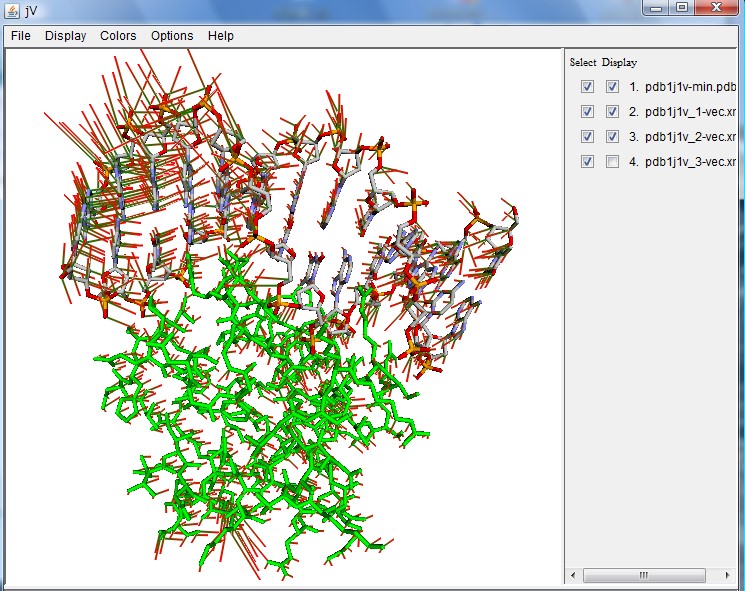
Mode 1、2、3を読み込み、Mode 1、2、3を同時に表示している。
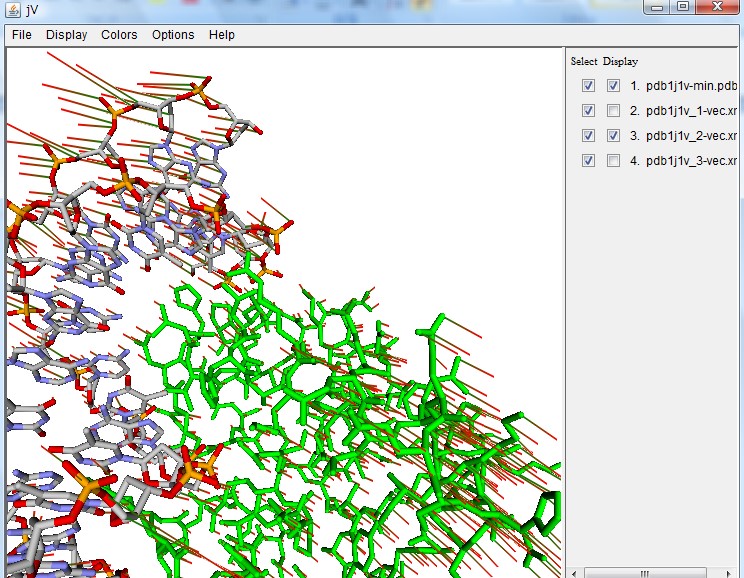
Mode 2のみを表示。
2.10 xxxx_n-vec.Jmol-script
モード番号 n の基準振動の変位ベクトルを Jmol で描画するためのデータ。
[jmolで変位ベクトルを描画する]
① Jmol を起動し、対応するPDBデータ(xxxx-min.pdb)を読み込む。 [File] → [Open]
② 変異ベクトル描画用ファイルを読み込む。 [File] → [Open]
(ここまで、表示された分子の並進・回転・拡大等の操作はしないこと)。
各変位ベクトルは2本の線分からなり、$line##A***, $line##B***
と名づけられている。##はモード番号であり(したがって、## = 01, 02, ...,
10)、***の部分は変位ベクトルの通し番号である。そこで、複数のモードの変位ベクトルを読み込んだ場合、Jmol の
console画面([File] → [Output console ...])で,
hide $line07*
などと打ち込めば、特定のモード(この場合はモード番号7)を非表示にでき、
display $line05*
などと打ち込めば、特定のモード(この場合はモード番号5)を表示することができる。
2.11 xxxx-note.txt
計算に関するコメント。
●立体構造の描画、アニメーション表示のためのソフト
jV ダウンロード、マニュアル等は こ
ちら から
Jmol ダウンロード、マニュアル等は こ
ちら から