GWASが可能になったのは、次世代シーケンサーが登場し、安価で高速にDNAの塩基配列が読み取れるようになったからです。そこで、次世代シーケンサーのポイントとな る点を説明しておきましょう。
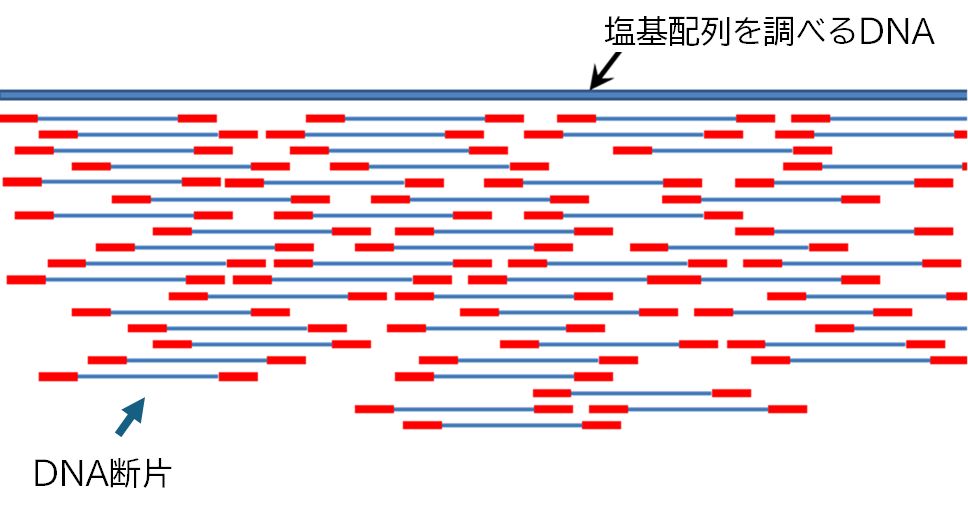
塩基配列を読み取りたいDNAは、まずランダムに細かく断片にし、それぞれの断片ごとに塩基配列を解読します。DNAの切断には、制限酵素とよばれる酵素 が使 われます。制限酵素は複数種類あり、それぞれDNAの特定の配列を認識して、そこでDNAを切断するため、同じDNAから異なった断片群を作り出すことができ ます。
次に、切断されたDNA断片の塩基配列の解読は、DNAをいったん1本鎖にした後、再び複製伸長させて行うのですが、そのとき本来のヌクレオチドである dNTP(デオキシヌクレオチド;dGTP、dATP、dCTP、dTTP がある)だけでなく、それとは少し異なるddNTP(ジデオキシヌクレオチド;ddGTP、 ddATP、 ddCTP、 ddTTP がある)を人為的に混ぜておきます。すると、複製伸長するとき、ある確率でその偽物のヌクレオチドを取り込むことが起こります。しかし、たとえば dNTP の代わりに ddNTP が取り込まれると、そこでDNA鎖の伸長が停止してしまいます。このことを利用してDNAの塩基配列が調べられます。
たとえば、DNA断片とともにdGTPとddGTPの両方を混ぜておくと、DNA断片を鋳型として、CのところにdGTPあるいはddGTPのどちらかが 結 合す るのですが、ddGTPが結合した場合には、複製はそこで止まってしまいます。そのため、同じDNA断片に対してさまざまな長さのDNAが複製されてくること になります。したがって、こうして複製されてきたDNAの長さを調べると、鋳型DNAのCの位置がわかるという仕組みです。
簡単な例で説明しましょう。元の配列が GTCTGAAACA であるとします。ジデオキシヌクレオチドを1種類だけまぜたDNA合成系を用意すると、次のようなDNA断片が生じることになります。[ ] 内の数字はその断片の塩基数です。
dTTP と ddTTP を使った場合、 [6] GTCTGA、[7] GTCTGAA、[8] GTCTGAAA、[10] GTCTGAAACA
dATP と ddATP を使った場合、 [2] GT、[4] GTCT
dCTP と ddCTP を使った場合、 [1] G、[5] GTCTG
dGTP と ddGTP を使った場合、 [3] GTC、[9] GTCTGAAAC
これらをその長さ順に並べることができますので、そうすると
G
T
A
C
[1] G
[2] GT
[3] GTC
[4] GTCT
[5] GTCTG
[6] GTCTGA
[7] GTCTGAA
[8] GTCTGAAA
[9] GTCTGAAAC
[10] GTCTGAAACA
となり、塩基配列GTCTGAAACAを確定することができま す。
ところで、各断片の塩基配列がわかっても、これでは、どの断片とどの断片がつながっていたという情報を得ることができません。そこで、複数の制限酵素を使ってさまざまな 断片を作 り、塩基配列が重なる領域を探し出し、その情報からもともとの塩基配列を推定します。これにはコンピュータの力を借ります。いわゆる”生物学”のイメージとは異なる生命へ のアプローチがあることを感じていただけたでしょうか。
